Биоинформатический анализ данных NGS
В последнее время, кроме известных и вошедших в стандартную обработку данных NGS программного конвейера Cell Ranger и R-пакета Seurat, появились дополнительные методы обработки. В нашем институте мы решаем самые актуальные задачи интерпретации полученных данных и используем самые последние подходы к анализу данных RNA-seq и scRNA-seq, а также методы машинного обучения, в частности — нейронные сети.
Типирование клеток
 |
 |
 |
 |
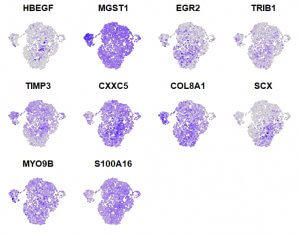
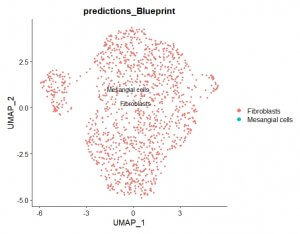
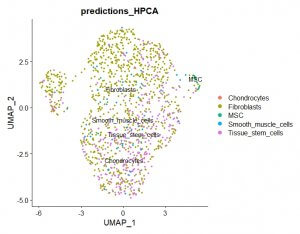
На сегодняшний день используется типирование клеток по специфическими маркерами. Как правило, задача сводится к выявлению дифференциально экспрессирующихся генов и их сравнению с такими известными клеточными референсами, как Cell Marker и PanglaoDB. Однако, многие ученые отмечают, что такой подход к типированию актуален больше для конечно дифференцированных форм клеток. Известно, что в любой популяции, доля клеток, завершивших свой путь дифференцировки незначительная. Остальные клетки представляют собой переходные формы, находящиеся на пути от исходной формы к конечной.
С этой точки зрения, типирование по биологическим процессам оказывается более информативным, так как если не дает точной информации о типе клетки, то помогает заглянуть внутрь и посмотреть, какие процессы для клетки были актуальны на момент секвенирования.
Еще одним подходом к типированию клеток может быть использование знаний о дифферонах клеток и предположение типа клеток на основании положения кластера клеток с неизвестным типом на линии, между кластерами клеток с известными типами.
RNA-velocity
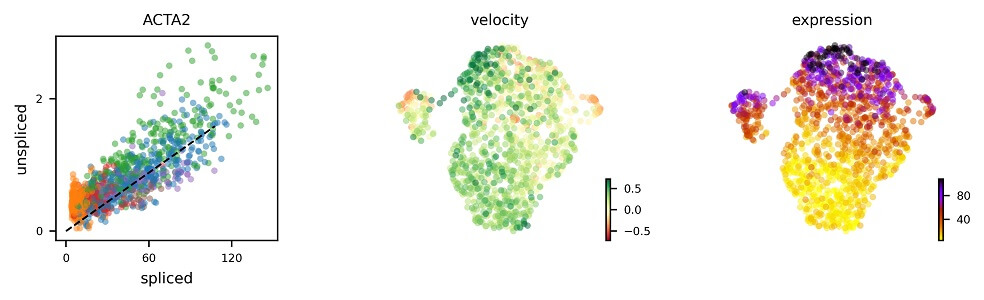
Попыткой изучить происходящие в клетке процессы не только в момент секвенирования является RNA velocity, производное от уровня экспрессии гена, оцениваемое разницей между сплайсированными и несплайсированными формами мРНК.Количество РНК это значимый индикатор состояния клетки. С помощью scRNA-seq у нас появилась возможность изучать РНК клетки с высокой точностью, чувствительностью и производительностью. Однако, этот метод позволяет сделать лишь статический «снимок» транскриптома клетки в момент времени, что лишает нас возможности ответить на вопросы, связанные, например, с эмбриогенезом и регенерацией, так как это динамические процессы.
Метод RNA-velocity в сочетании с пакетом scVelo позволяет расположить клетки образца в линию и «пронаблюдать» изменения уровня экспрессии генов в псевдовремени. Помимо привычной в стандартных анализах информации об уровне экспрессии, применяя этот метод, мы также получаем информацию о транскрипционной динамике и можем получить список «переходных» генов, играющих ключевую роль в последовательных состояниях клетки в процессе дифференцировки.
Траектории развития
 |
 |
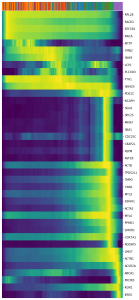 |
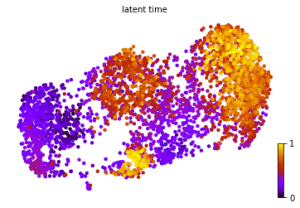
Предложенный метод скорости РНК позволил восстановить направление транскрипционной динамики, используя тот факт, что недавно транскрибированные, несплайсированные пре-мРНК, обнаруживаемые по интронным участкам и зрелые, сплайсированные мРНК можно различить в протоколе scRNA-seq.
Chromium Single Cell Immune Profiling
Chromium Single Cell Immune Profiling — это набор инструментов для анализа и визуализации иммунного репертуара совместно с данными об уровне экспрессии генов и определенными для конкретного эксперимента антителами. В результате такого анализа у исследователя появляется возможность посмотреть распределение аллельных вариантов (например, доминантный, рецессивный и гетерозиготный генотип), выбранных антител и уровня экспрессии генов.

